Enolase
L' enolasi , nota anche con i nomi di fosfopiruvato idratasi o 2-fosfoglicerato disidratasi , è un metalloenzima responsabile della catalizzazione che converte il 2-fosfo D- glicerato (2PG) da fosfoenolpiruvato (PEP), il 9 ° e ultimo passaggio preliminare del glicolisi :

|
H 2 O + |

|
| 2-fosfoglicerato | Fosfoenolpiruvato |
Enolase appartiene alla classe delle liasi . L'enolasi può anche catalizzare la reazione inversa, a seconda delle concentrazioni dei substrati nel mezzo. Il pH ottimale per questo enzima è 6,5. L'enolasi è presente in tutti i tessuti e organismi in grado di svolgere glicolisi o fermentazione . L'enzima è stato scoperto da Lohmann e Meyerhof nel 1934 e da allora è stato isolato da vari organi di origine come il muscolo umano e gli eritrociti .
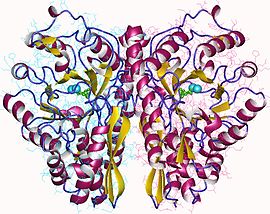
Fosfopiruvato idratasi Enolasi dimerica di lievito complessata con 2PG e PEP ( PDB 2ONE ).
Enolasi dimerica di lievito complessata con 2PG e PEP ( PDB 2ONE ).
| CE n. | CE |
|---|---|
| numero CAS | |
| Cofattore / i | Magnesio |
| IUBMB | Voce IUBMB |
|---|---|
| IntEnz | Vista IntEnz |
| BRENDA | Ingresso BRENDA |
| KEGG | Ingresso KEGG |
| MetaCyc | Passaggio metabolico |
| PRIAM | Profilo |
| PDB | Strutture |
| PARTIRE | AmiGO / EGO |
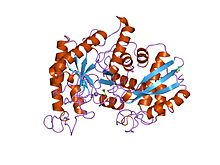 Enolase Homarus gammarus ( PDB 1PDZ )
Enolase Homarus gammarus ( PDB 1PDZ )
| Pfam | PF03952 |
|---|---|
| Clan Pfam | CL0227 |
| InterPro | IPR020811 |
| PROSITÀ | PDOC00148 |
| SCOP | 1els |
| SUPERFAMIGLIA | 1els |
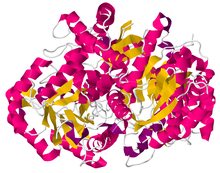 Enolasi 3 umana ( PDB 2XSX )
Enolasi 3 umana ( PDB 2XSX )
| Pfam | PF00113 |
|---|---|
| InterPro | IPR000941 |
| PROSITÀ | PDOC00148 |
Isoenzimi
L'enolasi ha tre subunità, α, ß e γ, ciascuna codificata da un gene diverso che può combinarsi per formare cinque diversi isoenzimi : αα, αß, αγ, ββ e γγ. Tre di questi isoenzimi (tutti omodimeri) si trovano più frequentemente nelle cellule umane adulte rispetto agli altri:
- αα, o enolasi non neuronale (NNE), si trova in tutti i tipi di tessuti come fegato, cervello, reni, milza
e tessuto adiposo. È anche chiamato enolase 1 - ββ o enolasi muscolo-specifica (MSE). Conosciuto anche come enolasi 3.
- γγ o enolasi specifica del neurone (NSE). Chiamato anche enolase 2 .
Struttura
L'enolasi ha un peso molecolare compreso tra 82.000 e 100.000 dalton a seconda della sua isoforma . Nell'alfa enolasi umana, le due subunità sono antiparalleli orientate in modo tale che il Glu 20 di una subunità forma un legame ionico con Arg 414 dell'altra subunità. Ogni subunità ha due domini distinti. Il piccolo dominio N-terminale è costituito da tre eliche alfa e quattro fogli beta . Il grande dominio C-terminale inizia con due fogli β seguiti da due eliche α e termina con una cavità circondata da fogli β alternati ad eliche α disposte a circondare i fogli β. La struttura compatta e globulare dell'enzima risulta da significative interazioni idrofobiche tra questi due domini.
Enolase è un enzima molto ben custodito con cinque siti attivi particolarmente importanti per l'attività. Rispetto al tipo selvaggio enolasi - enolasi che differisce da Glu mutato 168 , Glu 211 , Lys 345 o Lys 396 residuo - ha un livello di attività che è diminuita di un fattore di 105. Analogamente In questo modo, le variazioni sua 159 lasciano l'enolasi mutata con solo lo 0,01% della sua attività catalitica. Una funzione essenziale dell'enolasi è regolata dai suoi due cofattori Mg 2+ nel sito attivo, che servono a stabilizzare le cariche negative del substrato.
-
Struttura 3D di un dimero di enolasi con orientamento antiparallelo. Glu 20 N-terminale di un dimerico forma un legame ionico con Arg 414 C-terminale dell'altro per stabilizzare la struttura quaternaria dell'enzima.
-
Sito attivo di enolasi.
Meccanismo
Utilizzando sonde isotopiche, il meccanismo completo per la conversione di 2-PG in PEP è una reazione di eliminazione di tipo E1cb che coinvolge un carbanione intermedio. Il meccanismo dettagliato che segue si basa su studi cinetici cristallografici e chimici. Quando il substrato, 2-fosfoglicerato, si lega all'α-enolasi, il suo gruppo carbossilico forma un legame di coordinamento con i due ioni magnesio nel sito attivo. Questo stabilizza la carica negativa dell'ossigeno deprotonato aumentando l'acidità dell'idrogeno alfa. Il Lys 345 dell'enolasi deprotona l'idrogeno alfa e la carica negativa risultante viene stabilizzata sull'ossigeno del carbossilato dalla risonanza del cofattore ione magnesio. Dopo la formazione del carbanione intermedio , l'idrossido in C3 viene eliminato con acqua grazie a Glu 211 , e si forma così il PEP.
Inoltre, all'interno dell'enzima possono verificarsi cambiamenti conformazionali che favoriscono la catalisi. Nell'α-enolasi umana, il substrato è orientato nella posizione che favorisce il legame dell'enzima dovuto alle interazioni con i due ioni catalitici del magnesio, Gln 167 e Lys 396 . I movimenti in loop del Ser 36 verso His 43 , del Ser 158 verso il Gly 162 e dell'Asp 255 verso l' Asn 256 permettono al Ser 39 di legarsi al Mg 2+ e racchiudere così il sito attivo. Oltre a questo coordinamento con gli ioni magnesio catalitico, il pKa del substrato alfa idrogeno diminuisce a causa della protonazione del gruppo fosforile da parte di His 159 e della sua vicinanza ad Arg 374 . Arg 374 è anche responsabile della deprotonazione di Lys 345 nel sito attivo, conferendogli così il suo ruolo essenziale nel meccanismo.
Usi diagnostici
In recenti esperimenti medici, le concentrazioni di enolasi sono state campionate allo scopo di diagnosticare alcune condizioni neoplastiche e la loro gravità. Ad esempio, concentrazioni più elevate di enolasi nel liquido cerebrospinale (CSF) erano maggiormente correlate alla formazione di astrocitomi maligni rispetto ad altri enzimi testati (aldolasi, piruvato chinasi, creatina chinasi e lattato deidrogenasi). Lo stesso studio ha mostrato che il tumore progrediva più rapidamente nei pazienti con una maggiore concentrazione di enolasi nel liquido cerebrospinale. Sono stati identificati livelli aumentati di enolasi anche in pazienti che hanno recentemente sofferto di infarto miocardico o ictus. I livelli di enolasi nel liquido cerebrospinale, NSE sierica (tipo γγ) e creatinchinasi (tipo ßß) sono stati dedotti come indicativi per la valutazione della prognosi nelle vittime di arresto cardiaco. Altri studi si sono concentrati sul valore prognostico delle quantità di NSE nelle vittime di ictus. Altri studi si sono concentrati sul valore prognostico delle concentrazioni di NSE nelle vittime di ictus.
Gli autoanticorpi diretti contro la nostra alfa-enolasi sono associati a una malattia rara che è l'encefalopatia di Hashimoto.
Inibizione del fluoro
Il fluoruro è un noto concorrente per il substrato 2-PG da attaccare all'enolasi. Il fluoro fa parte di un complesso con magnesio e fosfato, che si lega al sito attivo dell'enolasi invece del 2-PG. In questo modo, bere acqua arricchita di fluoro fornisce fluoro a un livello che inibisce l'attività dei batteri enolasi nella bocca senza nuocere alla salute della persona. L'interruzione della via glicolitica dei batteri - e quindi la sua normale funzione metabolica - previene la formazione della carie dentale.
Note e riferimenti
- (in) Todd Larsen, Joseph E. Wedekind, Ivan Rayment e George H. Reed , " Un carbossilato del substrato di ossigeno fa da ponte tra gli ioni di magnesio nel sito attivo dell'enolasi: struttura del lievito enzimatico complessato con la miscela di equilibrio di 2 -Fosfoglicerato e fosfoenolpiruvato a 1.8 Å Risoluzione " , Biochemistry , vol. 35, n o 14, 9 aprile 1996, p. 4349-4358 ( PMID 8605183 , DOI 10.1021 / bi952859c , leggi in linea )
- I valori per la massa e il numero di residui qui indicati sono quelli del precursore della proteina risultante dalla traduzione del gene , prima delle modifiche post-traduzionali , e possono differire significativamente dal valori corrispondenti per la proteina funzionale.
- (it) Pancholi V, “ α-enolasi multifunzionale: il suo ruolo nelle malattie ” , Cell Mol Life Sci. , vol. 58, n o 7,Giugno 2001, p. 902–20 ( PMID 11497239 , DOI 10.1007 / PL00000910 , leggi in linea )
- (en) Hoorn RK, Flickweert JP, Staal GE, " Purificazione e proprietà dell'enolasi degli eritrociti umani " , Int J Biochem , vol. 5, n os 11-12,1974, p. 845–52 ( DOI 10.1016 / 0020-711X (74) 90119-0 )
- (de) Lohman K & Meyerhof O (1934), Über die enzymatische umwandlung von phosphoglyzerinsäure in brenztraubensäure und phosphorsäure ( Trasformazione enzimatica dell'acido fosfoglicerico in acido piruvico e fosforico ). Biochem Z 273, 60-72.
- (en) Peshavaria M, Day IN, " Struttura molecolare del gene dell'enolasi muscolo-specifico umano (ENO3) " , Biochem J. , vol. 275 (Pt 2), n o Pt 2,Aprile 1991, p. 427–33 ( PMID 1840492 , PMCID 1150071 )
- (in) Dinovo EC Boyer PD, " Sonde isotopiche del meccanismo di reazione dell'enolasi " , J Biol Chem , vol. 240,1971, p. 4586–93
- (in) Poyner RR Laughlin LT, Sowa GA, GH Reed, " Verso l'identificazione di catalizzatori acido / base nel sito attivo dell'enolasi: confronto delle proprietà delle varianti K345A, E168Q e E211Q " , Biochemistry , vol. 35, n o 5,Febbraio 1996, p. 1692–9 ( PMID 8634301 , DOI 10.1021 / bi952186y )
- (in) Reed GH Poyner RR Larsen TM, I Wedekind, Rayment I, " Studi strutturali e meccanicistici dell'enolasi " , Curr Opin Struct Biol. , vol. 6, n o 6,Dicembre 1996, p. 736–43 ( PMID 8994873 , DOI 10.1016 / S0959-440X (96) 80002-9 , leggi in linea )
- (in) Wedekind JE, Reed GH Rayment I, " Coordinazione ottaedrica nel sito di metallo ad alta affinità en enolasi: analisi cristallografica del complesso enzimatico MgII da lievito a risoluzione 1.9 Å " , Biochemistry , vol. 34, n o 13,Aprile 1995, p. 4325–30 ( PMID 7703246 , DOI 10.1021 / bi00013a022 )
- (in) Wedekind I, Poyner RR Reed GH Rayment I, " La chelazione della serina 39 a Mg 2+ blocca un cancello nel sito attivo dell'enolasi: struttura del complesso bis (Mg 2+) dell'enolasi di lievito e l'analogo intermedio fosfonoacetoidrossammato a risoluzione 2.1-Å " , Biochemistry , vol. 33, n o 31,Agosto 1994, p. 9333–42 ( PMID 8049235 , DOI 10.1021 / bi00197a038 )
- (in) Larsen TM Wedekind I, Rayment I, Reed GH " Un ossigeno carbossilato del substrato collega lo ione magnesio nel sito web attivo dell'enolasi: struttura dell'enzima di lievito complessata con la miscela di equilibrio di 2-fosfoglicerato e fosfoenolpiruvato a 1,8 Å resolution " , Biochemistry , vol. 35, n o 14,Aprile 1996, p. 4349–58 ( PMID 8605183 , DOI 10.1021 / bi952859c )
- (en) Duquerroy S, C Camus, J Janin, " Struttura a raggi X e meccanismo catalitico dell'enolasi dell'aragosta " , Biochemistry , vol. 34, n o 39,Ottobre 1995, p. 12513–23 ( PMID 7547999 , DOI 10.1021 / bi00039a005 )
- (in) Royds JA, Timperley WR, Taylor CB, "I livelli di enolasi e altri enzimi nel liquido cerebrospinale hanno evidenza di cambiamenti patologici " , J Neurol Neurosurg Psychiatr. , vol. 44, n o 12,Dicembre 1981, p. 1129–35 ( PMID 7334408 , PMCID 491233 , DOI 10.1136 / jnnp.44.12.1129 )
- (it) Roine RO, Somer H, Kaste M, Viinikka L, Karonen SL, " Esito neurologico dopo arresto cardiaco extraospedaliero. Previsione mediante analisi enzimatica del liquido cerebrospinale ” , Arch Neurol. , vol. 46, n o 7,Luglio 1989, p. 753–6 ( PMID 2742544 )
- (it) Hay E, Royds JA, Davies Jones GA, Lewtas NA, Timperley WR, Taylor CB, " Cerebrospinal fluid enolase in stroke " , J Neurol Neurosurg Psychiatr. , vol. 47, n o 7,Luglio 1984, p. 724–9 ( PMID 6747647 , PMCID 1027902 , DOI 10.1136 / jnnp.47.7.724 )
- (it) Fujii A, Yoneda M, Ito T, Yamamura O, Satomi S, Higa H, Kimura A, Suzuki M, Yamashita M, Yuasa T, Suzuki H, Kuriyama H, " Autoanticorpi contro il terminale amminico dell'alfa-enolasi sono un utile marker diagnostico dell'encefalopatia di Hashimoto ” , J. Neuroimmunol. , vol. 162, n osso 1-2,Maggio 2005, p. 130–6 ( PMID 15833368 , DOI 10.1016 / j.jneuroim.2005.02.004 )
- (in) Centers for Disease Control, "Le popolazioni che ricevono inviano acqua potabile pubblica fluorurata in modo ottimale Single-United States, 2000 " , MMWR Morb Mortal Wkly Rep. , vol. 51, n o 7,Febbraio 2002, p. 144–7 ( PMID 11905481 )
- (in) Hüther FJ, Psarros N, H Duschner, " Isolamento, caratterizzazione e cinetica di inibizione dell'enolasi da Streptococcus rattus FA-1 " , Infect Immun. , vol. 58, n o 4,1 ° aprile 1990, p. 1043–7 ( PMID 2318530 , PMCID 258580 , leggi in linea )
- (fr) Questo articolo è parzialmente o interamente tratto dall'articolo di Wikipedia in inglese intitolato " Enolase " ( vedi la lista degli autori ) .